本站讯(记者 李天畅 通讯员 杨治庆)近日,海南大学万逸研究员团队和清华大学李景虹院士团队共同在《Angew. Chem. Int. Ed.》发表论文:靶RNA激活CRISPR/Cas14a1进行反式切割单链DNA而靶RNA自身不被降解。海南大学为第一单位,海南大学博士研究生韦阳道和副研究员杨治庆为文章共同第一作者。
RNA是生物体重要的遗传物质,其碱基突变与各种疾病的发生密切相关,因此对早期生物体内RNA碱基突变进行诊断至关重要。目前RNA碱基突变主要采用有荧光定量PCR、高通量测序、CRISPR/Cas酶法等检测方法,由于CRISPR/Cas系统具有顺式和反式切割的能力,可实现多种病原体的高灵敏度、定量、快速检测,因此被广泛应用于RNA诊断。
当前用于RNA碱基突变诊断多为CRISPR/Cas13a系统,其他CRISPR/Cas系统多需依赖结合其他方法进行。因此,急需探索其他可直接用于区分RNA碱基突变的CRISPR/Cas系统。


据悉,研究以激活底物靶ssDNA做对照,设计靶RNA激活CRISRP-Cas14a1反式切割的生化实验,包括靶RNA的长度及缺失,sgRNA靶向片段的长度及靶RNA单碱基突变等一系列试验。研究发现:靶RNA(20 nt)激活Cas14a1反式切割活性最好,3’端相比5’端缺失碱基对Cas14a1酶的反式切割活性影响更大;且Cas14a1区分靶RNA单碱基错配能力要优于靶ssDNA。该研究使CRISPR/Cas14a1系统具有诊断RNA单碱基错配的潜力,为开发RNA单碱基诊断和成像技术奠定理论基础。
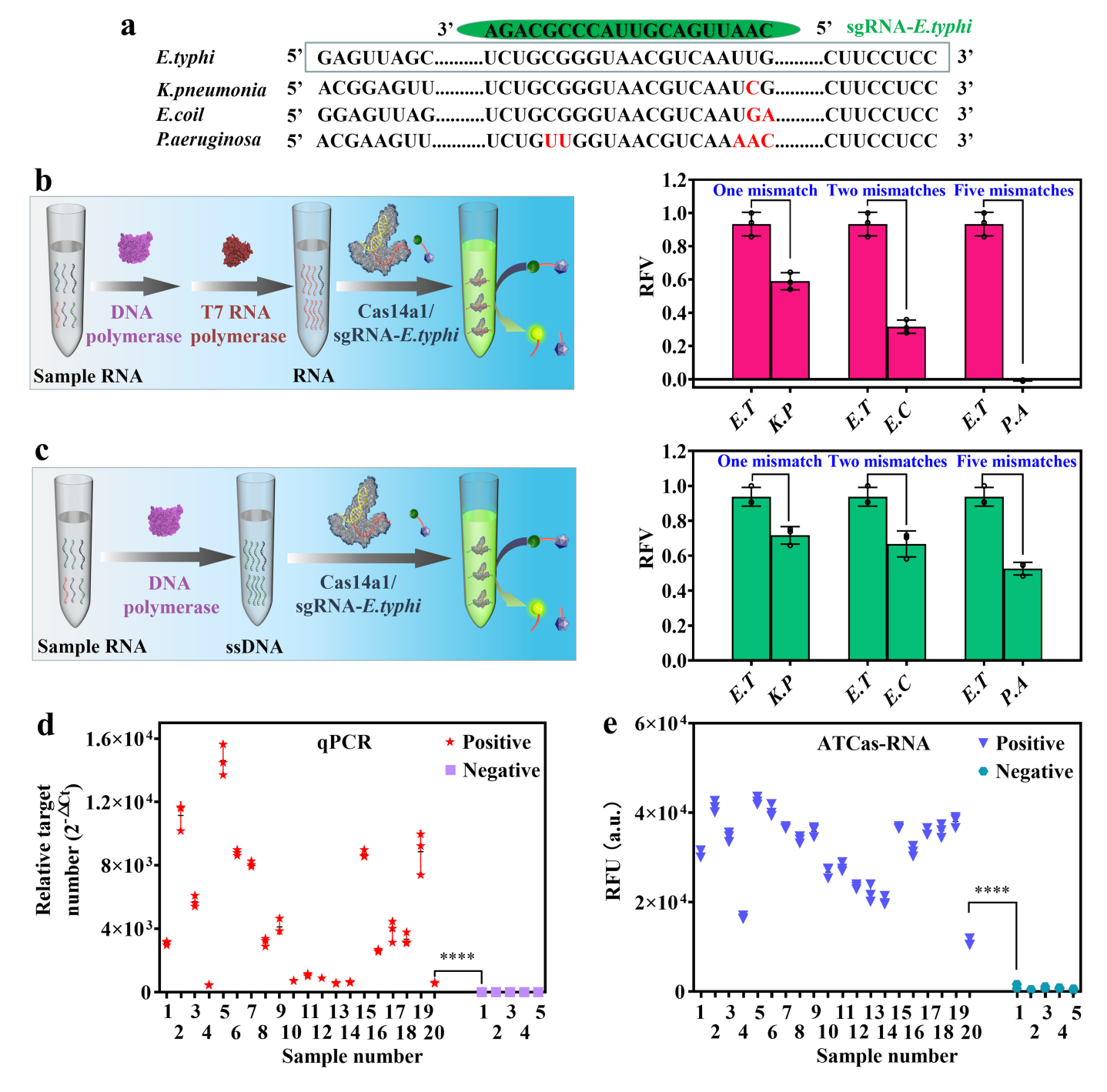
作者利用靶RNA激活Cas14a1引发反式切割功能开发出ATCas-RNA分析平台,其对检测16s RNA 基因序列相近的病原微生物具有良好鉴定能力。研究发现对25份实际样本检测的准确率达到100%。该技术的出现将有助于开发新的RNA分析方法,有望成为一种强大的RNA临床诊断工具。
论文信息:trans Single-Stranded DNA Cleavage via CRISPR/Cas14a1 Activated by Target RNA without Destruction
链接:
https://onlinelibrary.wiley.com/doi/full/10.1002/ange.202110384
(编辑:余梦月)
 公安部备案号:46010802000190 Copyright © 2024 hainan university
公安部备案号:46010802000190 Copyright © 2024 hainan university




